中国疾控中心在《新英格兰医学杂志》上的论文到底讲了什么?
时间:2020-02-13 点击: 次 来源:凤凰网 作者:钛媒体 - 小 + 大
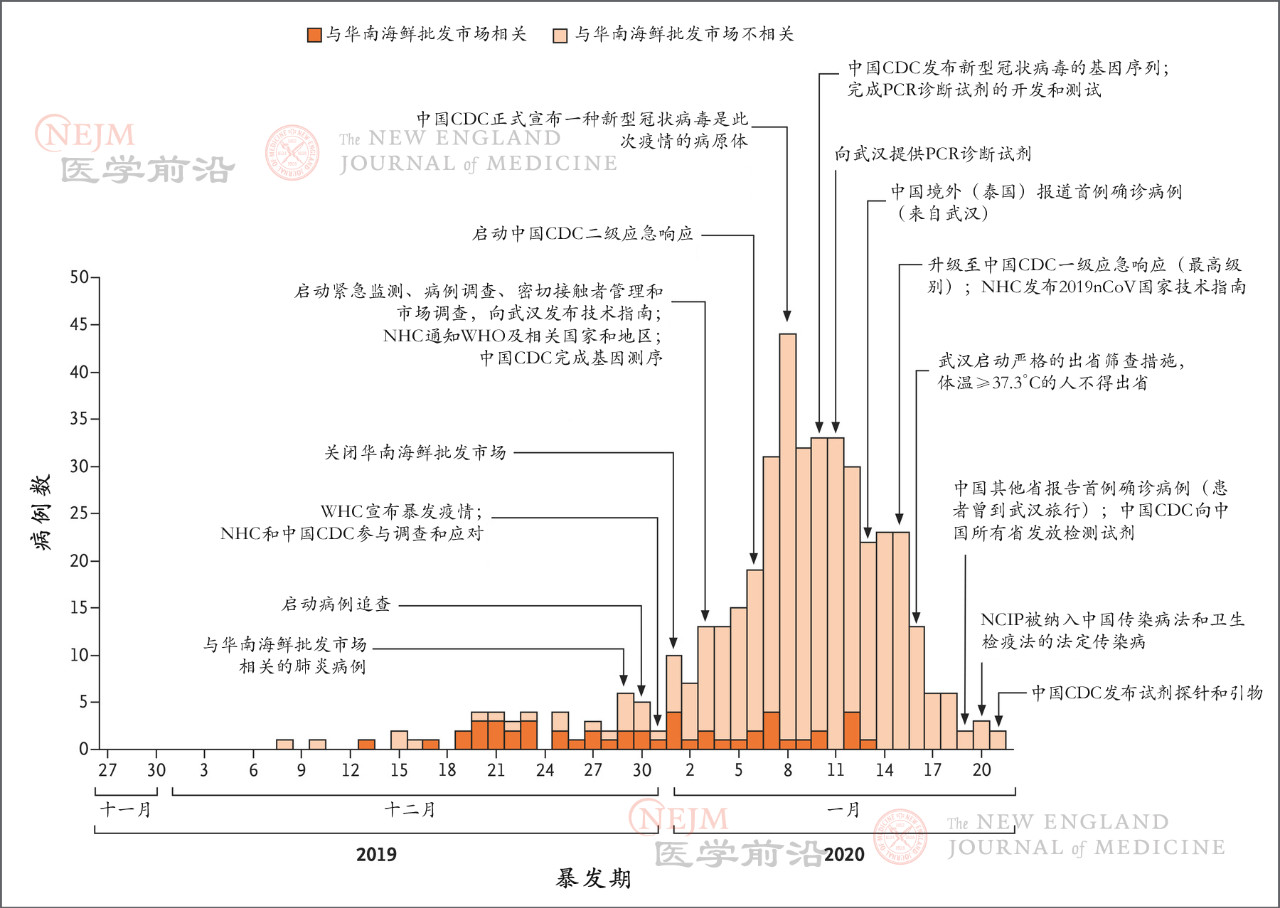
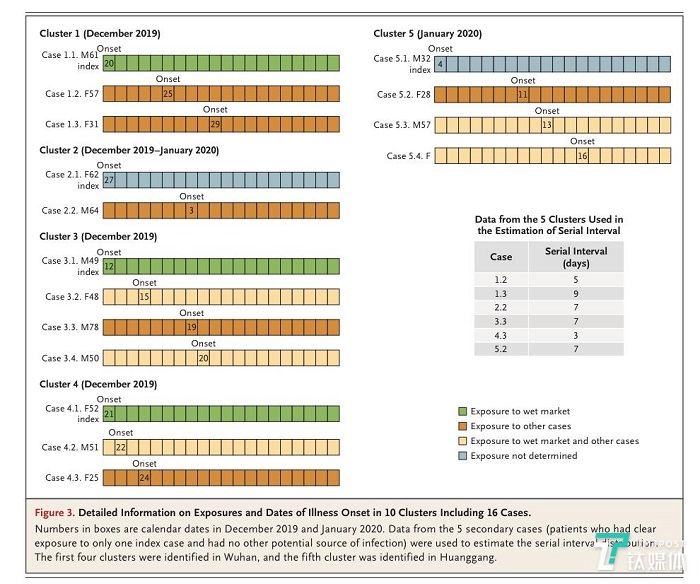
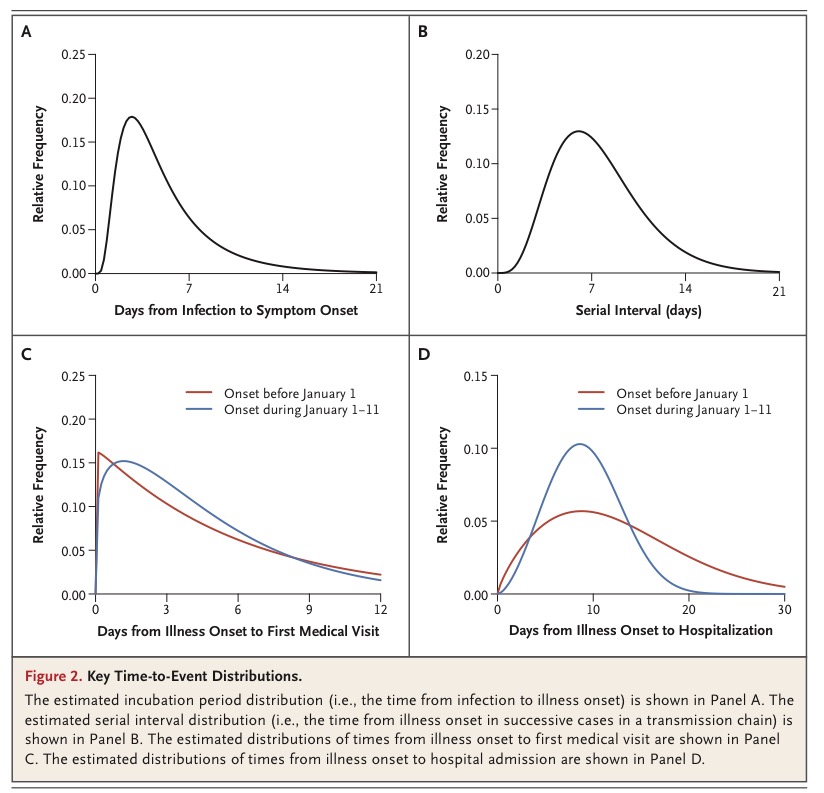
北京时间1 月 30 日,世界著名医学期刊《新英格兰医学杂志》发表最新文章,以“新型冠状病毒感染的肺炎在中国武汉早期传播动力学”为题,通过 425 例新冠肺炎确诊患者数据,首次披露截至 1 月 22 日新冠病毒传播的规律。 整理后的论文作者信息明细图 据悉,本篇论文是由中国疾病控制预防中心(下称中国疾控中心,CDC)、湖北省疾控中心、香港大学李嘉诚医学院公共卫生学院等多单位共同完成。经过钛媒体整理后发现,在这份作者名单当中,除了中国疾控中心主任高福、中国疾控中心副主任冯子健、湖北省疾控中心主任杨波等人,还包括澳门大学中药质量研究国家重点实验室教授赵静教授、香港前行政长官办公室主任,香港首任食物及卫生局副局长,现在是香港大学李嘉诚医学院院长的梁卓伟等两岸三地的科学家,可谓是首次非常权威、最为详细的新冠病毒早期流行病学数据披露。 值得一提的是,在本篇论文提到,事实上,在 2019 年 12 月中旬,新冠病毒在密切接触者之间就已发生人际传播,也就是所谓的“人传人”。此外,根据论文中的说法,在 2020 年 1 月 11 日前,已经有 7 例武汉医护人员感染发病。 这一消息引起广泛传播,中国疾控中心就被推到了风口浪尖,网友纷纷质疑,是不是 12 月中旬发生新冠病毒“人传人”之后就没有公布,自己却抢着发论文? 对此,1 月 31 日,中国疾控中心作出回应「请阅读中国疾控中心回应发表论文:所有病例在论文撰写前已向社会公布」 一位知情人士对八点健闻表示,该论文的研究和发表,滞后于最初对病毒的理解,也滞后于信息有限情况下得出的决策。CDC 不太可能在已知病毒人传人的情况下,在防控措施上做出不符合这一判断的决策。 “中国疾控中心早期的防控决策,是基于对病毒发展阶段的判断,是按照冠状病毒发展的第一阶段特征——动物传人的理解上进行的,忽略了实际上新冠已进展到人传人的第二阶段。” 尽管对此回应大家评价褒贬不一,但对这篇发表在《新英格兰医学杂志》的论文所提到的各种数据不失为一个让大众更好了解早期新冠病毒传播规律的窗口,钛媒体也据此复盘了整个研究过程以及早期传播检测的步骤问题。 如何检测与鉴定?科学判断与调查数据是一方面 数据检测 本篇论文提到,新冠肺炎最早的病例是通过“病因不明的肺炎”监视机制鉴定的。首先,最初发病的 4 名病患,被定义为一种没有病因病原体的疾病,医生认为,这些病患满足以下标准: 1、发烧(≥38°C) 2、肺炎的影像学证据 3、遵循标准临床指南进行抗菌治疗 3 到 5 天后,并且无症状改善 4、比正常肺炎的白细胞或淋巴细胞计数低 为了快速识别肺炎病例,并努力提高早期发现的敏感性,于 2020 年 1 月 3 日,疾控中心制定了量身定制的监测方案,包括以上的判断方法,使得之后的病患可以尽快查找出来病因,以及确定潜在的新冠肺炎病例。 而在发现疑似病例之后,由中国疾控中心成员,省、市、县级别的地方疾病预防控制中心组成了联合现场流行病学团队,在收到通知之后启动详细的现场调查,并收集呼吸道标本,以便在中国疾控中心病毒研究所进行集中检测,接着,由中国疾控中心和地方疾控中心工作人员组成的联合团队,对所有疑似和确诊的 2019-nCoV(新型冠状病毒)病例进行了详细的现场调查,确定传染和感染的疑似病例。 通过采访受感染者,亲戚,近亲和医护人员等密切接触者,将数据收集到标准化表格中。值得一提的是,研究人员在必要时,会与每位感染患者及其亲属进行了访谈,发生突变前两周内的接触史,包括接触野生动物的日期,时间,频率和方式,尤其是否与华南海鲜批发市场有关联或接触,暴露于任何相关环境下。而且,研究人员也会去调查,包括有关与具有类似症状的病患接触到的信息,患者发病前 2 周内曾去过的家庭和地方等。 让中国疾控中心的联合团队,可以最快定位病患群体,控制传播源和切断传播途径,减少传播率。 在调查中收集的所有流行病学信息,包括接触史,事件时间表和紧密联系者身份,所有流行病学信息都与多个来源的信息进行了交叉核对。数据一式两份输入中央数据库,并通过 EpiData 软件(EpiData Association)进行验证。 病例定义 针对感染不明原因肺炎(NCIP)的疑似病例,根据世界卫生组织(WHO)在 2003 年和 2012 年的建议,将基于非典 SARS 和中东呼吸综合征 MERS 的案例进行处理和解决病例问题。 值得一提的是,定义未知肺炎病例需要满足以下四个标准: 1、发烧,有无体温记录; 2、肺炎的放射学证据; 3、白细胞计数低或正常或淋巴细胞计数低; 4、按照标准临床指南进行抗菌治疗3天后症状没有减轻 符合上述全部四条标准的肺炎病例,或者符合前三条标准,并且与华南海鲜批发市场有流行病学接触或关联,以及接触过其他类似症状患者的肺炎病例,可以定义为 NCIP 病例,并采取个案变范围性的疑似肺炎病例案件,也就是俗称的“群体性”病例。 而根据论文中叙述,2020年1月18 日,一旦获得了已确诊病例的新信息之后,更新了用于定义这一疑似病例的流行病学新标准: 在发病前 14 天内出现发烧或出现呼吸道症状,并曾到武汉旅行,或与来自武汉人(患者)有密切接触者。 对于确诊病例的定义为,通过以下三种方法中的一个,并在呼吸道标本中监测到新型冠状病毒(2019-nCoV)呈阳性,也就是李兰娟院士所说的“咽拭子”和试剂盒检测方案,才可以确诊: 1、分离出2019-nCoV; 2、实时逆转录聚合酶链反应(RT-PCR)至少有两次显示2019-nCoV阳性; 3、检测出与2019-nCoV匹配的基因序列。 实验室检测 新型冠状病毒实验室测试分析,基于 WHO 先前的建议,取病患的上、下呼吸道标本。提取 RNA 标本,并使用由武汉病毒研究所所研制出的 2019-nCoV 特异性引物和探针,进行确诊,并通过实时 RTPCR 进行测试。 测试是在湖北省疾控中心的生物安全中心设施中进行的,然后在中国疾控中心的国家病毒疾病控制研究所进行再审核。 如果两个靶标通过特定的实时 RT-PCR 检测为阳性,则认为该病例已在实验室确认。将小于 37 的循环阈值(Ct 值)定义为阳性,将大于等于 40 的 Ct 值定义为阴性。Ct 值定义为 37 至 40 的,定义为中等载量,需要通过重新测试进行确认。 如果复测之后,Ct 值小于 40,并且观察到明显的峰值,则重新检测为阳性。 通过三种方法之一,从患者的支气管肺泡灌洗液样本中鉴定出基因组:Sanger 测序、Illumina 测序或纳米孔测序。在中国疾病预防控制中心增强的生物安全实验室设施中,将呼吸道标本接种到细胞中进行病毒分离。 统计分析 根据发病日期绘制流行曲线,覆盖与流行病识别和控制有关措施的关键日期,以帮助解释这个病因的缘由。描述病例特征,包括人口统计学特征, 以及暴露的卫生保健工作者的状况。通过对数正态分布,拟合潜伏期分布(即从感染到发病的时间延迟)以及暴露史、发病日期的数据(在一部分病例中具有可用的详细信息),将首次就诊和入院就诊的病患信息,呈现 Weibull 分布拟合,并对来自联合团队的聚集类调查数据,进行整合与拟合,呈现伽马分布,和估计序列间隔分布,重新具体定义,传播链中连续病例中疾病发作日期之间的延迟,来估计并确定新冠病毒的传播力与传播范围。 另外,这一统计图表,是分析 12 月 10 日至 1 月 4 日期间发病病例的数据来估算疫情的增长速度,预计在 12 月 13 日武汉疫情正式宣布之后,发现的感染病例将很快增加,带有人畜共患病感染的发热日期模型(使用更新方程式来计 算)来确定与华南海产品批发市场是否有发病关联,并且我们使用此模型来得出流行病的增长率,流行病的倍增时间和基本繁殖数( R0)。 基于 SARS 期间的 R0 数值,即平均值为 8.4,标准差为 3.8,来判断新冠病毒的传播是否比 SARS 更强,毒性是否更强烈。 疾控中心使用大数据,即MATLAB 软件(MathWorks)对潜伏期, 序列间隔,生长速率和 R0 进行分析。使用 SAS 软件(SAS Institute)和 R 软件(R Foundation for Statistics Computing)进行了具体分析。 数据结果:老年男性患者更易感染新冠病毒 根据论文中解释,流行病的发生是跟着病例呈指数增长,最初几天的下降很可能是由于对最近发作的病例的不确定性以及鉴定和报告的延迟,而不是发病率的真正拐点。 论文提到的数据,显示12月就发生了人际之间传播 非常重要的是,上图曲线的后半部分并不表示事件案例数量有所减少,而是由于截止日期之前确 定案例的时间推迟了。考虑到随着时间的推移,检测试剂盒的可用性和使用量会增加, 因此在解释 1 月份病例增长速度时应格外小心。 通过对武汉市截至 1月22日,共425例新冠肺炎确诊患者进行分析发现,他们中位年龄为 59 岁(范围为 15 至 89 岁),56% 为男性,即 240 例。15 岁以下的儿童没有早期病例。总的来说,老年男性患者更易感染新冠病毒,而且更有可能在华南海鲜批发市场有过接触。 患新冠肺炎的病人特征数据统计 作者团队还在论文当中,对截至目前的病例时间段做了分析,称三个早期传播阶段: 第一个阶段是针对 1 月 1 日(即华南海鲜批发市场关闭)之前发病的患者; 第二个阶段是发病时间为 1 月 1 日至 1 月 11 日的患者,这是向武汉提供 RT-PCR 试剂的日期。第三个阶段是那些在 1 月 12 日或之后发病的人。 在这三个时期中,医护人员中的病例比例逐渐增加。 并且在本论文的图表,以及摘要结论中表明,自 2019 年12月中旬以来,密切接触者之间已发生人际传播。根据论文中的一张图表,大多数最早的病例均报告了华南海鲜批发市场暴露史,但从12月底开始,与华南海鲜市场不相关的病例便呈指数增长。“总之,我们发现武汉现阶段的新冠肺炎病例倍增时间约为 7.4 天。密切接触者之间的人际传播从 12 月中旬开始已经发生,并在此后一个月内逐渐播散。” 关于病患暴露数据分析,论文提到,科研人员检查 10 例确诊病例的暴露数据,估计平均潜伏期为 5.2 天(95% 置信区间【CI】4.1 到 7.0)分布的第 95 个百分位是 12.5 天(95% CI,9.2 至 18),并且通过获得的 5 个病例组信息对发病时间确定,估计病毒序列间隔分布的平均值(±SD)为 7.5±3.4 天(95%CI,5.3 至 19)。 有关10个类群(包括16个病例)的暴露和疾病发作日期的数据信息 简单来说,就是新冠病毒的潜伏期,是在平均 10 天左右,也根据之前中国疾控中心最新的说法,潜伏期最早在3天,最迟在14天,而根据印度快报引述该国卫生部的说法,这一潜伏间隔时间,处理在 28天左右,防止再次传播。 在截至 2020 年 1 月 4 日的流行曲线中,传染病增长率为每天 0.10(95% CI,0.050~0.16),倍增时间为 7.4 天(95% CI,4.2~14)。应用上述由一人传至另一人的间隔时间分布情况,估计 R0 为 2.2(95% CI,1.4~3.9)。 在1月1日之前发病的 45 例患者中,从发病至首次就诊的平均间隔期估计为 5.8 天(95% CI,4.3~7.5),与1 月 1 日和 1 月 11 日之间的 207 名发病的患者相似。平均为 4.6 天(95%CI,4.1 至 5.1)左右。从而说明,尽管疫情被广泛传播,并且进行防控措施,但发病的患者并没有加快治疗力度。 关键疫情发生的时间分布曲线统计 在1月1日之前发病的 44 例患者中,从发病至住院的平均间隔期估计为 12.5 天(95% CI,10.3到~14.8),这一间隔期超过1月1日至1月11日期间发病的189例患者(均值,9.1天;95% CI,8.6~9.7)。 作者团队称,在图表当中,没有为 1 月 12 日或之后发病的患者绘制这些分布图,因为尚未发现那些近期发病且病程较长的患者。 据悉,在这份数据报告中,中国疾控中心、香港大学等作者团队成员,提供了 NCIP 传播动力学和流行病学特征的初步评估,并将数据首次公之于众。 更多人认为,早期传播模式通过数据展示给世人,对接下来全球对新冠肺炎的防控工作,利大于弊。 论文地址: https://www.nejm.org/doi/pdf/10.1056/NEJMoa2001316?articleTools=true 本论文里提到的引述信息: 1、The 2019-nCoV Outbreak Joint Field Epidemiology Investigation Team, Li Q. Notes from the field: an outbreak of NCIP (2019-nCoV) infection in China — Wu- han, Hubei Province, 2019–2020. China CDC Weekly 2020;2:79-80. 2. Tan WJ, Zhao X, Ma XJ, et al. A novel coronavirus genome identified in a clus- ter of pneumonia cases — Wuhan, China 2019–2020. China CDC Weekly 2020;2:61-2. 3. ZhuN,ZhangD,WangW,etal.A novel coronavirus from patients with pneumonia in China, 2019. N Engl J Med 2020 January 24 (Epub ahead of print). 4. Xiang N, Havers F, Chen T, et al. Use of national pneumonia surveillance to de- scribe inf luenza A(H7N9) virus epidemi- ology, China, 2004–2013. Emerg Infect Dis 2013;19:1784-90. 5. Munster VJ, Koopmans M, van Doremalen N, van Riel D, de Wit E. A novel coronavirus emerging in China — key questions for impact assessment. N Engl J Med. DOI: 10.1056/NEJMp2000929. 6. WHO guidelines for the global sur- veillance of severe acute respiratory syn- drome (SARS). 2004 (https://w w w.who .int/csr/resources/publications/WHO _CDS_CSR_ARO_2004_1.pdf?ua=1). 7. Middle East respiratory syndrome case definition for reporting to WHO. 2017 (https://www.who.int/csr/disease/ coronavirus_infections/mers-interim-case -definition.pdf?ua=1). 8. Azhar EI, El-Kafrawy SA, Farraj SA, et al. Evidence for camel-to-human trans- mission of MERS coronavirus. N Engl J Med 2014;370:2499-505. 9. New coronavirus pneumonia prevention and control program (2nd ed.) (in Chinese). 2020 (http://www.nhc.gov.cn/jkj/s3577/ 202001/c67cfe29ecf1470e8c7fc47d3b751e88 .shtml). 10. Laboratory diagnostics for novel coro- navirus. WHO 2020 (https://www.who.int/ health-topics/coronavirus/laboratory -diagnostics-for-novel-coronavirus). 11. Lipsitch M, Cohen T, Cooper B, et al. Transmission dynamics and control of severe acute respiratory syndrome. Sci- ence 2003;300:1966-70. 12. Bauch CT, Lloyd-Smith JO, Coffee MP, Galvani AP. Dynamically modeling SARS and other newly emerging respiratory ill- nesses: past, present, and future. Epide- miology 2005;16:791-801. 13. Paules CI, Marston HD, Fauci AS. Coronavirus infections — more than just the common cold. JAMA 2020 January 23 (Epub ahead of print). 14. Perlman S. Another decade, another coronavirus. N Engl J Med. DOI: 10.1056/ NEJMe2001126. 15. de Wit E, van Doremalen N, Falzarano D, Munster VJ. SARS and MERS: recent insights into emerging coronaviruses. Nat Rev Microbiol 2016;14:523-34. 16. WongG,LiuW,LiuY,ZhouB,BiY, Gao GF. MERS, SARS, and Ebola: the role of super-spreaders in infectious disease. Cell Host Microbe 2015;18:398-401. 17. Chan JF, Yuan S, Kok KH, et al. A fa- milial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster. Lancet 2020 January 24 (Epub ahead of print). 18. Wu P, Hao X, Lau EHY, et al. Real- time tentative assessment of the epide- miological characteristics of novel coro- navirus infections in Wuhan, China, as at January 2020. Eurosurveillance 2020; 25(3):pii=2000044. 19. Lipsitch M, Hayden FG, Cowling BJ, Leung GM. How to maintain surveillance for novel influenza A H1N1 when there are too many cases to count. Lancet 2009; 374:1209-11. |